无需原生质体,南科大开发植物单细胞核全长RNA检测技术
以下内容转载自南方科技大学生科院
近年来,高通量单细胞转录组测序已广泛应用于动物细胞研究,但只在少数植物物种和组织的研究中存在报道。其主要原因是因为植物细胞比动物多了一层细胞壁,使得植物单细胞RNA-seq必须先消化细胞壁并制备原生质体。而原生质体的制备对于许多组织都非常困难,且制备过程会影响细胞的转录状态而引入不必要的干扰。因此,一种无需原生质体化的方法对于单细胞测序技术在植物领域中更广泛的应用必不可少。
近日,南方科技大学生命科学学院翟继先团队,基于纳米孔测序技术,开发了一种不经过原生质体直接检测植物单细胞核全长转录组测序方法,相关成果以“FlsnRNA-seq: protoplasting-free full-lengthsingle-nucleus RNA profiling in plants”为题发表在基因组学领域知名学术杂志《Genome biology》上。
DOI:https://doi.org/10.1186/s13059-021-02288-0
根据翟继先课题组之前对新生RNA全基因组水平的研究发现,细胞核中有大量已经多聚腺苷酸化的信使RNA依然与染色质相结合。那么细胞核中的RNA能否表征整个细胞的转录状态呢?此外,基于短读长平台的单细胞测序往往只能捕获基因的表达丰度,那么与全长转录组测序技术相结合的单细胞全长测序能否利用RNA剪接、可变多聚腺苷酸化等导致的同源异构体信息来辅助细胞类型鉴定并挖掘出更多信息呢?
为了回答这些问题,并突破原生质体的限制,优化植物单细胞测序技术,翟继先团队提出了基于细胞核测序的全长单细胞转录组测序方法FlsnRNA-seq(protoplasting-free full-length single-nucleusRNA profiling in plants):通过结合10xGenomics单细胞测序技术和三代全长Nanopore测序方法实现了单细胞核全长转录组测序。同时利用短读长测序技术捕获表达丰度信息,并以Nanopore测序技术捕获同源异构体信息,从而最大限度地保留单个细胞核的转录状态(图1)。
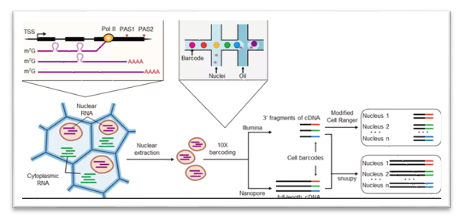
图1.FlsnRNA-seq流程图。首先,通过研磨的方法从组织样本中分离出细胞核,通过10x Genomics建库方案构建RNA文库,获得全长cDNA并进行扩增后,将DNA分成两半。一半用于常规的二代文库构建并Illumina测序,另一半用于Nanopore长片段测序。
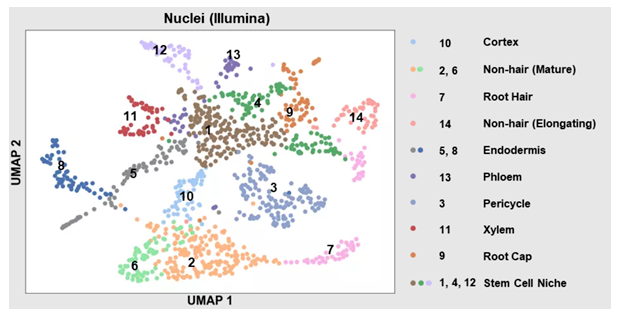
图2.FlsnRNA-seq可以捕获拟南芥根尖组织中的多种细胞类型。利用UMAP对单细胞核测序所获得的基因表达丰度信息降维并可视化。细胞的颜色代表不同的细胞簇。
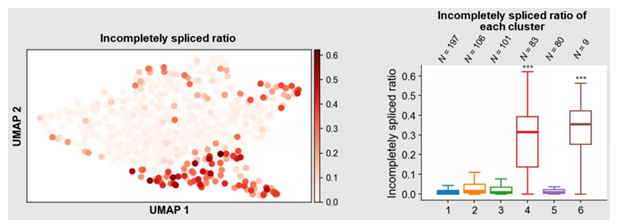
图3.FlsnRNA-seq可以识别拟南芥胚乳中不同簇中内含子剪切的差异。(左图)利用UMAP对单细胞核测序所获得的基因表达丰度信息降维并可视化。细胞的颜色由Nanopore测序技术所获得的未完全剪接的转录本的比例所决定。(右图)细胞的未完全剪接转录本比例在不同细胞簇中的分布情况。
由于植物细胞壁聚合物和细胞大小的巨大差异,植物单细胞转录组测序极具挑战性。基于纳米孔测序技术,作者开发了FlsnRNA-seq方法:对植物单细胞核进行全长转录组测序,在单细胞水平上检测剪接状态和可变多聚腺苷酸化,获得细胞基因转录和剪接信息,从而进一步提高细胞类型鉴定,且可以得到传统测序技术难以捕获的同源异构体信息。这充分说明纳米孔cDNA全长测序在识别,分类和发现罕见单细胞类型上的巨大潜力。
翟继先教授为该论文的通讯作者,南科大为唯一通讯单位。翟继先课题组研究助理教授龙艳萍博士、博士生刘智剑和研究助理教授贾津布博士为该论文共同第一作者,南科大生科院陈炜教授和研究副教授方亮博士也合作参与了该研究工作。该研究得到了科技部国家重点研发计划、广东省创新创业团队、深圳市科创委以及广东省植物细胞工厂分子设计重点实验室基金的资助。








